研究 (Research)
最終更新日:
時間を考慮したシングルセルデータ比較解析アルゴリズムの開発 (Software development in single-cell RNA data analysis considering pseudotime )
准教授 加藤 有己(医学系研究科 神経遺伝子学) KATO Yuki (Graduate School of Medicine)
研究の概要
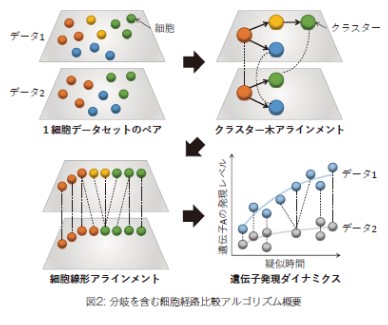
生体組織などの細胞集団の遺伝子発現情報を1細胞単位で表すシングルセルデータを用いて、細胞動態を考慮した細胞経路解析を実施することが可能である。特に、細胞動態に分岐を含む細胞経路比較は、解析者が事前知識を使って比較すべき線形経路を選択する必要があり、コンピュータによる自動化は実現されていなかった。今回我々は、計算機科学の分野で提案されていた木のアラインメント技術を応用することにより、細胞経路木に対して比較解析を行う手法 CAPITAL を開発した。これにより、比較すべき最適な経路がアルゴリズムにより決定され、細胞経路比較解析の精度向上を実現した。
研究の背景と結果
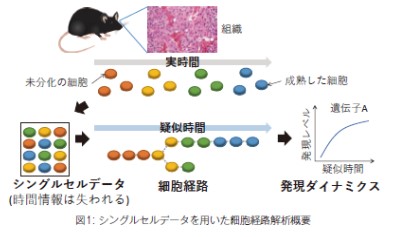
ある時刻で生体組織から取得した細胞集団は、未分化のものから分化したものまで、様々な経過時間の細胞を含むスナップショットであるとみなすことができる。このような不均質な細胞集団を、シングルセル発現データに基づいてクラスタリングし、対応する時系列を予測して並べることで、疑似的な時間軸に沿った細胞状態の遷移過程 (細胞経路) を捉えることが可能となる。これは特に細胞の採取回数が少なくて済むため、時間やコストの点でメリットがあり、「木を見て森を見る」アプローチとも言える。細胞経路解析の応用例として、2つの実験系のデータから導出される細胞経路をアラインメントすることで、条件の違いによって変化する制御遺伝子を同定できる。
本研究では、遺伝子発現が類似している細胞型を要素とした、2つの木構造の経路をあらかじめ推定し、それらをアラインメントした後に、個別の線形経路のペアにて細胞間の対応関係を計算するアルゴリズムCAPITAL (comparative analysis of pseudotime trajectory inference with tree alignment) を開発した。計算機実験では、分岐が複数箇所出現するように設計された合成データセットのペアを用いて、既存のデータ統合手法と網羅的に比較したところ、CAPITAL が統計的に有意に経路 (時系列) 情報を保存してアラインメントできることを示した。次に、異なる研究で得られたヒトの骨髄細胞の遺伝子発現データのペアに対し CAPITAL を適用したところ、異なるデータ間でも互いに同じ細胞型へのアラインメントに成功し、代表的なマーカー遺伝子の発現ダイナミクスが類似していることを確認した。さらに、ヒトとマウスから得られた骨髄細胞データに対して CAPITAL による比較解析を行い、異種間での遺伝子発現パターンの特徴やその差異を評価することに成功した。
研究の意義と将来展望
本研究成果により、組織や器官における細胞動態を1細胞レベルで比較する上で、複雑な細胞経路を効率良く扱うことが可能となった。例えば、血液など複雑に分化する細胞集団を考え、一方のデータとしてある疾患患者の、他方のデータとして健常者の遺伝子発現データを取得し、CAPITAL を実行することで、これまで検出できなかった疾患関連遺伝子の同定につながると考えている。現在はトランスクリプトームのみならず、エピゲノムやプロテオームなど、マルチオミクスデータを駆使して病態解明を目指す時代に入った。CAPITAL の計算原理はマルチオミクスデータを扱うことが可能であり、今後医学生物学研究に貢献できることが期待される。
担当研究者
准教授 加藤 有己(医学系研究科 神経遺伝子学)
キーワード
シングルセル/遺伝子発現/経路推定/アラインメント
応用分野
医療・ヘルスケア
参考URL
https://www.med.osaka-u.ac.jp/pub/rna/ykato/research.html
https://researchmap.jp/read0207242
